
近日,复旦大学代谢与整合生物学研究院王冠琳课题组在Life Metabolism期刊在线发表了题为“Optimized upstream analytical workflow for single-nucleus transcriptomics in main metabolic tissues”的研究论文。该研究聚焦于单核转录组测序(snRNA-seq)在白色脂肪组织、骨骼肌、肝脏及下丘脑等代谢旺盛组织中的应用,建立了一套系统化的标准分析流程,显著提升了数据质量及下游细胞类型注释的准确性。

代谢稳态的维持依赖多组织的协同作用,尤其是脂肪组织、肌肉、肝脏以及下丘脑等关键器官。这些组织的细胞功能失调与肥胖、2型糖尿病及非酒精性脂肪性肝病等代谢相关疾病密切相关1。snRNA-seq技术通过提取细胞核,避免了对组织进行剧烈解离的步骤,特别适用于脂肪组织等结构脆弱或冷冻保存的样本,近年来在代谢研究中展现出巨大潜力2。然而,由于该技术存在环境RNA污染和双细胞混杂问题,极易在下游分析中造成错误聚类和细胞类型误判,从而影响生物学信号的准确解释。
为解决上述难题,王冠琳课题组以多个高代谢活性组织样本为研究对象,对snRNA-seq分析流程中的关键步骤进行了全面的系统评估。研究团队基于人和小鼠的67个样本(共计201,411个细胞核),建立了一套涵盖从原始FASTQ文件处理到下游注释分析的完整上游流程,包括表达矩阵生成、环境RNA去除、双细胞识别、质量控制、标准化、数据整合与基准评估等关键步骤。其中,CellBender工具被用于环境RNA去除,scDblFinder被用于双细胞识别,Seurat被用于后续表达矩阵质控与标准化,多个主流整合算法(如Seurat CCA、Harmony、rPCA、scVI与scANVI)则被用于多样本数据整合,并通过scIB框架进行系统性评价。
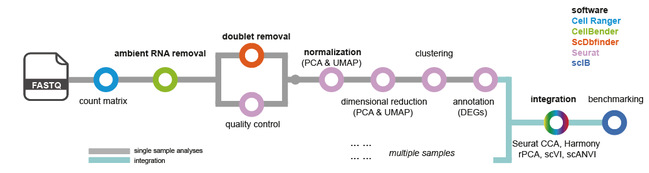
结果显示,环境RNA和双细胞污染在不同组织间存在显著差异,肝脏和白色脂肪组织中的背景RNA污染率高达90% ,而肌肉和下丘脑样本中表现出较高的双细胞比例。研究表明,未处理数据中脂肪细胞标志基因PLIN1、ADIPOQ与LPL呈弥散性表达,且不同细胞类型间存在显著混叠,严重干扰细胞分群。而经CellBender和scDblFinder处理后,PLIN1信号高度集中于脂肪细胞簇,细胞类型边界更加清晰,标志基因表达特异性大幅提升,类似结果在白色脂肪组织、肌肉、肝脏和下丘脑等多个组织中均得到验证。
在多样本整合分析中,研究团队评估了五种主流算法在批次效应校正与生物信号保持两方面的表现。综合scIB指标后发现,在分析的白色脂肪组织中,scANVI在保留细胞类型特异性方面表现最佳,而Harmony在多种组织中(如肝脏、肌肉、下丘脑)展现出更为均衡和稳定的整合效果,成为跨样本数据分析的优选方案。
综上,该研究强调了snRNA-seq数据在下游分析前进行严谨质量控制与预处理的重要性。通过优化环境RNA去除与双细胞识别步骤,不仅能够显著提高数据的整体质量,同时可以增强对真实细胞状态的识别能力,从而为深入解析代谢组织中的细胞构成及其功能提供了坚实基础。本研究还提供了一套可复用、可扩展的标准化分析流程,显著提升了分析准确性和可重复性,进一步拓展了单核转录组学在代谢疾病研究和精准医学中的应用前景。研究团队同时公开了完整的分析教程和代码(https://metabomicslab.github.io/snRNAseq-analysis-workflow/)。
本研究由复旦大学代谢与整合生物学研究院青年研究员王冠琳为本论文的通讯作者,博士研究生董鹏娓为第一作者,课题组博士研究生丁世彤也对本工作有重要贡献。研究工作得到了国家重点研发计划、国家自然科学基金、复旦大学启动基金等项目的支持。
原文链接:
https://doi.org/10.1093/lifemeta/loaf010
参考文献:
1.Silvestri, C., and Di Marzo, V. (2013). The endocannabinoid system in energy homeostasis and the etiopathology of metabolic disorders. Cell Metab. 17, 475-490.
2.De Donno, C., Hediyeh-Zadeh, S., Moinfar, A.A., Wagenstetter, M., Zappia, L., Lotfollahi, M., and Theis, F.J. (2023). Population-level integration of single-cell datasets enables multi-scale analysis across samples. Nat. Methods 20, 1683-1692.




 复旦大学网上办事服务大厅
复旦大学网上办事服务大厅 复旦大学实验室安全教育与管理平台
复旦大学实验室安全教育与管理平台