
代谢是一切生命活动的基础。代谢组学技术通过检测代谢物在生命体中的含量,反映出个体在基因与环境共同作用下的生化特征,尤其是新代谢物的发现将有助于探索生命活动的未知机制。譬如,促癌代谢物2-羟基戊二酸(2-hydroxyglutarate)的发现,为探索急性髓细胞白血病(AML)的癌变机理,及开发靶向抑制剂的癌症治疗方案奠定了基础1-3。代谢组学及多学科的交叉研究将促进发现与疾病相关的分子标志物,为疾病的早期发现和治疗提供重要的依据。
代谢组学近年来迅速发展,利用液相色谱-质谱技术可测出上千种不同的小分子代谢物,但是代谢组学的数据中仍有大量离子峰未被鉴定,意味着仍有潜在的代谢物待发现。如何全面的、准确的对这些离子峰进行标注,并发展新的分析手段,系统性的发现生命体中潜在新代谢物,也是代谢组学领域的前沿问题。分子网络分析(Molecular network analysis)利用离子峰之间的关系来扩大离子峰标注的广度并提高准确性,是近年来代谢组学数据分析的一个新趋势4-7。
2021年10月28日,复旦大学代谢与整合生物学研究院青年研究员陈立博士与美国普林斯顿大学Joshua D. Rabinowitz 课题组在Nature Methods发表了题为Metabolite discovery through global annotation of untargeted metabolomics data的文章。文章报道了名为NetID的新算法,可从非靶向代谢组学数据中系统的对代谢物进行标注,并用于发现未知代谢物。NetID软件目前已公开发布在Github网站(https://github.com/LiChenPU/NetID)。

NetID算法以已知的代谢组学信息为输入,以实验观察到的离子峰为网络节点,并以可以反映生物分子转化(如氧化还原反应,脱羧反应等)及质谱现象(如加和物、同位素等)的分子量差为边,将整体代谢组学数据拓展连接为分子网络。同时,本研究将网络分析算法中的全局网络优化的策略应用在代谢组学的分子网络分析中,生成可连接大多数观察到的离子峰的单一网络,充分利用所有离子峰的信息对整个网络进行全局最优化标注,从而提高峰标注的精度,并生成包含有用生物化学转化信息的离子峰之间的关系。该算法可广泛应用于代谢组学数据分析,从而对缺少MS2谱图的低丰度离子峰进行更好的标注,进而提高发掘潜在的新代谢物的覆盖范围和准确性。
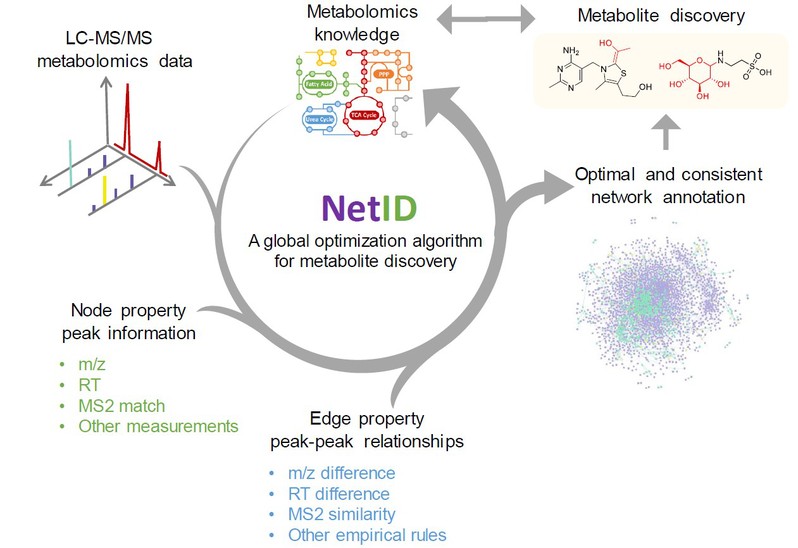
作者将NetID算法应用于酵母和小鼠的非靶向代谢组学数据中,对约3/4的非背景离子峰进行了标注,并成功鉴定出了五种未知代谢物,其中包括硫胺素衍生物和 N-葡萄糖基-牛磺酸。作者进一步通过同位素示踪的实验证实了小鼠体内的这些新代谢物的代谢过程。
综上,该研究的新算法NetID创新性的结合了代谢组学实验原理与网络优化算法,提出了全局化标注非靶向代谢组学数据的新策略,为发现和标注新代谢物有力的工具。
原文链接:
https://doi.org/10.1038/s41592-021-01303-3
参考文献:
Zhao, S. et al. Glioma-Derived Mutations in IDH1 Dominantly Inhibit IDH1 Catalytic Activity and Induce HIF-1α. Science 324, 261–265 (2009).
Dang, L. et al. Cancer-associated IDH1 mutations produce 2-hydroxyglutarate. Nature 462, 739 (2009).
DiNardo, C. D. et al. Durable Remissions with Ivosidenib in IDH1-Mutated Relapsed or Refractory AML. N. Engl. J. Med. 378, 2386–2398 (2018).
Shen, X. et al. Metabolic reaction network-based recursive metabolite annotation for untargeted metabolomics. Nature Communications 10, 1516 (2019).
Nothias, L.-F. et al. Feature-based molecular networking in the GNPS analysis environment. Nat Methods 17, 905–908 (2020).
Ludwig, M. et al. Database-independent molecular formula annotation using Gibbs sampling through ZODIAC. Nature Machine Intelligence 2, 629–641 (2020).
Dührkop, K. et al. Systematic classification of unknown metabolites using high-resolution fragmentation mass spectra. Nature Biotechnology 1–10 (2020) doi:10.1038/s41587-020-0740-8.




 复旦大学网上办事服务大厅
复旦大学网上办事服务大厅 复旦大学实验室安全教育与管理平台
复旦大学实验室安全教育与管理平台