
单细胞多组学技术是发现疾病潜在靶点的重要手段。2024年10月9日,我院王冠琳课题组与英国牛津大学Bethan Psaila, Adam Mead教授课题组合作在Science Translational Medicine在线发表题为“A proinflammatory stem cell niche drives myelofibrosis through a targetable galectin-1 axis”的研究论文,绘制了骨髓纤维化MPLW515L小鼠模型中各类骨髓细胞互作的单细胞图谱,揭示了Galectin-1(Gal-1)可以作为骨髓纤维化进展的生物标志物和治疗靶点,该研究也为理解骨髓纤维化疾病中造血细胞与其微环境相互作用提供了重要资源。
首先,研究团队详细分析了骨髓纤维化MPLW515L小鼠模型骨髓免疫细胞与基质细胞的组成。研究人员从对照组和MPLW515L小鼠的股骨、胫骨和髂骨中分别分离造血细胞和基质细胞,进行高通量单细胞转录组测序。通过质量控制、去除双细胞和批次效应校正后注释了包括基质细胞(mesenchymal stromal cells)、成纤维细胞(fibroblasts)、骨系细胞(osteolineage cells)、软骨细胞(chondrocytes)、内皮细胞在内的8种基质细胞,以及11种造血细胞类型,包括造血干/多能祖细胞(hematopoietic stem and multipotent progenitor cells)、巨核细胞(megakaryocytes)、T/B淋巴细胞、嗜酸/嗜碱/肥大细胞(eosinophil/ basophil/mast cells)、单核/巨噬细胞(monocytes/macrophages)和中性粒细胞(neutrophil)等,这是迄今为止最全面的骨髓纤维化中细胞和分子图谱。
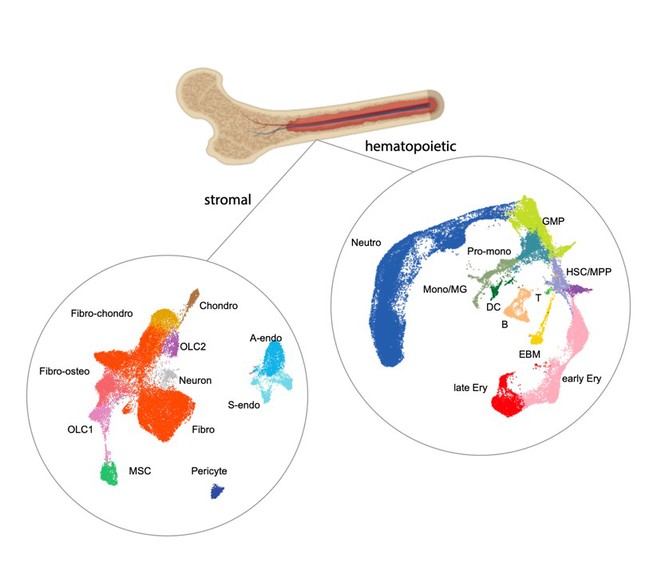
图1 骨髓纤维化单细胞图谱
接下来,研究团队深入探讨了骨髓纤维化中各细胞类型的相对丰度变化。与对照组相比,研究人员首次发现骨髓纤维化小鼠中嗜碱性粒细胞和肥大细胞(basophil, and mast cells)的丰度显著增加,同时还确认了基质细胞是骨髓中细胞外基质ECM的主要来源。由于成纤维细胞是最丰富的间质细胞类型,研究人员进一步鉴定了五个不同转录特征的成纤维细胞子集,发现了骨髓纤维化疾病富集和TNF-NFKB症途径显著高表达的炎性iFibs亚群。针对EBM细胞和iFibs亚群,研究团队通过推断细胞类型间的配体-受体相互作用以探讨其细胞间通信如何发生改变。结果显示,嗜碱性粒细胞、肥大细胞、巨核细胞与基质中的MSCs和iFibs的相互作用在骨髓纤维化中显著增强,表明这4种细胞是骨髓中炎症信号的重要“协调者”。
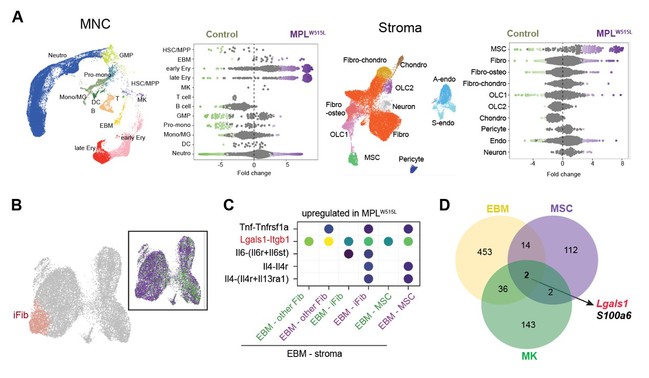
图2 骨髓纤维化疾病细胞组成和基因表达变化
特别地,研究人员在这些关键相互作用的细胞中发现了编码Gal-1蛋白的基因Lgals1相互作用信号的高表达,提示Gal-1 信号可能在骨髓纤维化的进展中发挥关键作用。通过结合患者队列,研究人员在人骨髓活检和外周血无创检测中也发现Gal-1 表达与纤维化的进展正相关,通过类器官和小鼠实验发现抑制Gal-1表达能够缓解纤维化水平以及小鼠脾脏肿大。这些结果都表明Gal-1作为髓系恶性肿瘤的关键病理介质,有希望作为疾病进展的生物标志物和潜在治疗靶点。

图3 Gal-1在骨髓纤维化病人中高表达
综上所述,本研究构建了骨髓纤维化的造血细胞与基质细胞图谱及其相互作用网络,揭示了肥大细胞、巨核细胞以及 iFibs 在骨髓纤维化的炎症和纤维化过程中的关键作用,发现了重要的生物标志物和治疗靶点—Galectin-1。该研究展示了单细胞多组学分析在发现和验证疾病新靶点中的应用。
复旦大学代谢与整合生物学研究院青年研究员王冠琳为本研究的共同第一作者及共同通讯作者,王冠琳课题组的博士研究生董鹏娓和实习生周文江同学参与了本研究的数据分析。
原文链接:
https://doi.org/10.1126/scitranslmed.adj7552
参考文献:
1.Khoury, J.D., Solary, E., Abla, O., Akkari, Y., Alaggio, R., Apperley, J.F., Bejar, R., Berti, E., Busque, L., and Chan, J.K. (2022). The 5th edition of the World Health Organization classification of haematolymphoid tumours: myeloid and histiocytic/dendritic neoplasms. Leukemia 36, 1703-1719.
2.Nangalia, J., and Green, A.R. (2017). Myeloproliferative neoplasms: from origins to outcomes. Hematology 2014, the American Society of Hematology Education Program Book 2017, 470-479.
3.Masarova, L., Bose, P., Pemmaraju, N., Daver, N.G., Sasaki, K., Chifotides, H.T., Zhou, L., Kantarjian, H.M., Estrov, Z., and Verstovsek, S. (2022). Improved survival of patients with myelofibrosis in the last decade: single‐center experience. Cancer 128, 1658-1665.
4.Jeremy Wen, Q., Yang, Q., Goldenson, B., Malinge, S., Lasho, T., Schneider, R.K., Breyfogle, L.J., Schultz, R., Gilles, L., and Koppikar, P. (2015). Targeting megakaryocytic-induced fibrosis in myeloproliferative neoplasms by AURKA inhibition. Nature Medicine 21, 1473-1480.
5.Malara, A., Gruppi, C., Abbonante, V., Cattaneo, D., De Marco, L., Massa, M., Iurlo, A., Gianelli, U., Balduini, C.L., and Tira, M.E. (2019). EDA fibronectin–TLR4 axis sustains megakaryocyte expansion and inflammation in bone marrow fibrosis. Journal of Experimental Medicine 216, 587-604.
6.Psaila, B., Wang, G., Rodriguez-Meira, A., Li, R., Heuston, E.F., Murphy, L., Yee, D., Hitchcock, I.S., Sousos, N., and O’Sullivan, J. (2020). Single-cell analyses reveal megakaryocyte-biased hematopoiesis in myelofibrosis and identify mutant clone-specific targets. Molecular Cell 78, 477-492. e478.




 复旦大学网上办事服务大厅
复旦大学网上办事服务大厅 复旦大学实验室安全教育与管理平台
复旦大学实验室安全教育与管理平台